Unsere analytischen Methoden sind modular aufgebaut und können nutzbringend kombiniert werden.
Wählen Sie eine unserer Serviceleistungen, um mehr über die Methode zu erfahren.
Unser Ziel ist es Ihre Fragen zu beantworten.
Die Polymerase Kettenreaktion (Englisch: polymerase chain reaction PCR) ist die grundlegendste und wichtigste Arbeitsmethode der IDENTXX.
Für die erfolgreiche Durchführung einer PCR braucht es vier Komponenten
Ablauf der PCR
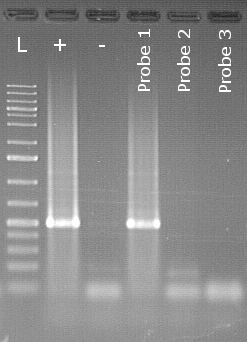
Für qualitative Nachweise, auch als End-Punkt-PCR bezeichnet, wird die Enzymreaktion zunächst komplett durchgeführt. Nach dem Ende der Reaktion werden die entstanden Produkte in einem Agarosegel der Größe nach aufgetrennt und mit Hilfe von Fluoreszenzfarbstoffen sichtbar gemacht.
Spezifischer Endpunkt-PCR-Nachweis von Peronospora belbahrii
Im Beispiel ist ein Agarosegel eines Nachweises von Peronospora belbahrii
dem Falschen Mehltau des Basilikums aus Saatgut dargestellt. Neben dem
Größenmarker (L) befindet sich die Positivkontrolle (+) gefolgt von der Negativkontrolle (-).
In der Spur der Positivkontrolle erkennt man einen deutlich weiß leuchtenden Bereich,
welcher auch als „Bande“ bezeichnet wird. Hierbei handelt es sich um das angefärbte spezifische PCR-Produkt in der Spur der Probe 1 ist ebenfalls eine Bande auf gleicher Höhe zu sehen. Die Proben wird daher als positiv bewertet. Die Probe 2 und 3 zeigen keine Banden und werden daher als negativ bewertet.
Unsere qualitativen PCR-Nachweise sind sicher und robust, und kommen beim Nachweis der unterschiedlichsten Pathogenen zum Einsatz.
Eine weitere wichtige PCR-Methode ist die quantitative PCR (qPCR), welche auch als Echtzeit-PCR bezeichnet wird. Sie folgt den gleichen Reaktionsprinzipien wie die qualitative PCR. Jedoch wird die Reaktionslösung um eine weitere Komponente, die sogenannte Sonde ergänzt. Diese befindet sich zwischen den Primern und ist ebenfalls spezifisch für den gesuchten DNA-Abschnitt.
Findet nun eine PCR-Reaktion statt, baut das Polymerase-Enzym die Sonde ab. Ein dabei freigesetzter Fluoreszenzfarbstoff (Fluorophor) kann währende diesen Abbauprozessen in Echtzeit detektiert werden. Je mehr PCR-Produkte entstehen, desto mehr Fluorophor wird freigesetzt. Der Thermocycler gibt diesese Messwerte in Form einer Kurve wieder. Je früher es zu einem exponentiellen Anstieg der Kurve kommt, desto mehr DNA war in der Ausganglösung vorhanden.
Die Nutzung von Sonden hat noch einen weiteren Vorteil. Durch die Verwendung von unterschiedlichen Fluorophoren können wir bis zu 5 PCR-Reaktionen in einem Reaktionsgefäß durchführen (Multiplex-Analysen). Dadurch ist es möglich, den Gehalt von spezifischer Pathogen-DNA (Im oberen Kurvenverlauf Blau dargestellt) ins Verhältnis zur DNA der Wirtspflanze zu setzten (Grün dargestellt).
Dieser Ansatz hat mehrere Vorteile:
Wir nutzten die Endpunkt-PCR auch als Basisreaktion für die DNA-Sequenzierung, hierbei unterscheiden wird zwischen der Sanger-Sequenzierung und der Pyrosequenzierung. Zwei wichtige Analysewerkzeuge, die wir für unterschiedliche analytische Zwecke einsetzten.
Diese Methode wurde von Frederick Sanger und Alan Coulson in der 1970ziger Jahren entwickelt und bildet neben der PCR-Reaktion das Fundament der modernen Molekularbiologie. Die Methode ist so einfach wie genial. Der fertigen Endpunkt-PCR-Reaktionslösung werden spezielle Oligonukleotide zugesetzt, welche mit Fluoreszenzfarbstoffen markiert sind. In einer anschließend durchgeführten PCR-Reaktionen erhält man DNA-Fragmente von unterschiedlicher Länge, welche am Ende jeweils ein fluorerszenzmarkiertes Oligonukleotid tragen, welche sich mit Hilfe entsprechender Detektoren auswerten lassen. Als Ergebnis erhält man eine Abfolge von farbigen Spitzen mit der entsprechenden Zuordnung zu den vier Basen der DNA.
Als Grundlage für die Pyrosequenzierung dient eine Endpunkt-PCR. Durch die Verwendung eines biotynilierten Primers trägt je ein Strang der entstehenden PCR-Produkte am Ende Biotin (labeling). Durch die kovalente Bindung an Streptavidin kann dieser gelabelte DNA-Strang abgetrennt und gereinigt werden. Während der Sequenzierreaktion werden getrennt voneinander die die vier Basen zur Reaktionslösung zu gegeben. Findet ein Einbau statt, wird ein enzymreaktions-basierter Lichtblitz frei, welcher wie bei der qPCR in Echtzeit detektiert wird.
Der entscheidende Punkt ist die Quantifizierung des Nukleotideinbaus. Dadurch kann der Grad der Zygotie einer TSR-Position exakt bestimmt werden.
Als Grundlage für die Pyrosequenzierung dient eine Endpunkt-PCR. Durch die Verwendung eines biotynilierten Primers trägt je ein Strang der entstehenden PCR-Produkte am Ende Biotin (labeling). Durch die kovalente Bindung an Streptavidin kann dieser der gelabelte DNA-Strang abgetrennt und gereinigt werden. Während der Sequenzierreaktion werden getrennt voneinander die vier Basen zur Reaktionslösung zu gegeben. Findet ein Einbau statt, wird ein enzymreaktions-basierter Lichtblitz frei, welcher wie bei der qPCR in Echtzeit detektiert wird.
Der entscheidende Punkt ist die Quantifizierung des Nukleotideinbaus. Dadurch kann der Grad der Zygotie einer TSR-Position exakt bestimmt werden.
Als Grundlage für die Fragmentlängenanalyse dient eine Endpunkt-PCR. Durch die Verwendung eines fluorophor-markierten Primers trägt je ein Strang der entstehenden PCR-Produkte am Ende einen Fluoreszenzfarbstoff (labeling).
Mit Hilfe der hochauflösenden Kapillarelektrophorese, welche mit einer Detektoreinheit für Fluoreszenzsignale ausgestattet ist, können die Längen der PCR-Fragmente auf das Nukleotid genau bestimmt werden.
Eine Fragmentlängenanalyse kommt bei folgenden Analysen zu Einsatz:
Um methylierte Cytosine zu identifizieren, muss die DNA noch vor der Endpunkt-PCR einer Bisulfit-Reaktion ausgesetzt werden. Bei dieser Reaktionen wird für alle Cytosine, welche nicht methyliert sind und direkt vor einer Guanin-Base (CpG-Dinukleotid) stehen, ein Uracil eingebaut.
Bei der anschließenden Endpunkt-PCR wird das Uracil durch Thymin ersetzt, wodurch eine im Vergleich zum Original abweichende Basenabfolge entsteht.
Die Sequenz kann nach dem Ende der Reaktion entweder nach der Sanger-Methode oder mit der Pyro-Methode bestimmt werden.
Eine Methylierungs-Analyse kommt bei folgenden Anwendungen zu Einsatz:
In Kooperation mit zwei führenden Einrichtungen bietet die IDENTXX Gewächshausbiotest zur Ermittlung der metabolischen Resistenz, auch als nTSR bezeichnet, an.
Die Partner verfügen sowohl über einen modernen Spritzstand als auch ausreichend Gewächshauskapazität und Fachpersonal.
Folgende Punkte sind hierbei besonders hervorzuheben:
Kontakt
Anschrift
Information